Señalización TOR y autofagia en microalgas
José L. Crespo y M. Esther Pérez-PérezEl objetivo principal de nuestra investigación es entender cómo las microalgas adaptan su crecimiento y metabolismo a las condiciones de estrés y a los desafíos medioambientales, utilizando para ello la microalga modelo Chlamydomonas reinhardtii y una nueva microalga extremófila, Chlamydomonas urium. Para ello, llevamos a cabo dos líneas de investigación:
- Estudiar la ruta de señalización TOR en respuesta a distintos estímulos nutricionales. IP: José L. Crespo.
- Investigar los mecanismos moleculares implicados en la regulación redox de la autofagia en respuesta a estrés. IP: M. Esther Pérez-Pérez.
TOR (Target Of Rapamycin) es un regulador central del crecimiento celular en todos los eucariotas que integra señales nutricionales y energéticas. Nuestros estudios previos han demostrado que la ruta de señalización TOR está estructural y funcionalmente conservada en C. reinhardtii (revisado en Mallén-Poce et al. J Exp Bot 2022), y regula procesos fundamentales como la traducción (Díaz-Troya et al. Plant Physiol 2011) y la autofagia (Pérez-Pérez et al. Plant Physiol 2010). Además, hemos mostrado que la actividad TOR responde a nutrientes esenciales como nitrógeno, fósforo y carbono. Nuestros resultados indican que los niveles de fósforo regulan la señalización TOR vía LST8, una subunidad conservada del complejo TOR 1 (TORC1) que interacciona con el dominio quinasa de TOR. La limitación de fósforo produce una notable disminución en los niveles de la proteína LST8, que finalmente resulta en una inhibición de la actividad TOR (Couso et al. Plant Cell 2020). Por otro lado, hemos demostrado recientemente que el carbono inorgánico es un regulador central de TOR, ya que la asimilación fotosintética de CO2 activa eficientemente a la ruta TOR a través de la síntesis de aminoácidos, particularmente Gln (Mallén-Ponce et al. PNAS 2022). Actualmente, estamos investigando los metabolitos específicos que conectan la disponibilidad de CO2 con TOR en respuesta a la luz. Así mismo, estamos analizando el papel de TOR en la biogénesis del cloroplasto en Chlamydomonas.
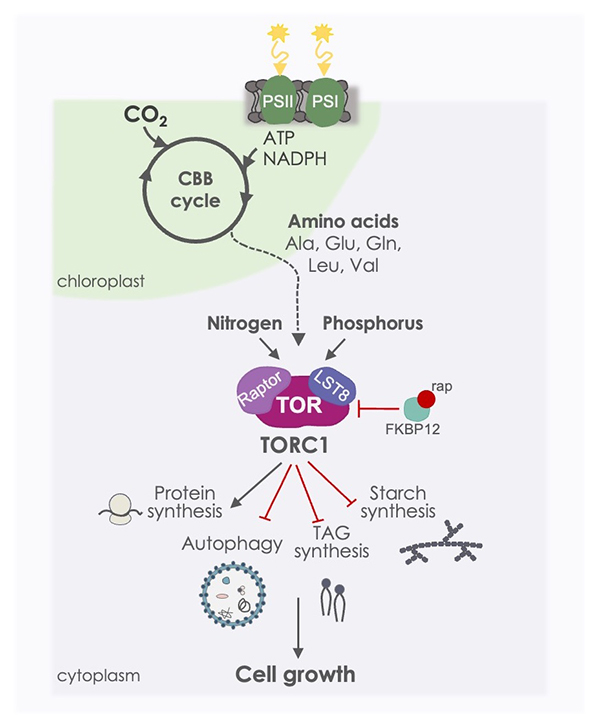
Figura 1. La ruta de señalización TOR en Chlamydomonas. TORC1 es un regulador central del crecimiento celular en Chlamydomonas, mediante la activación de procesos anabólicos como la síntesis de proteínas y la inhibición de procesos catabólicos como la autofagia. En microalgas, TOR también controla la síntesis de lípidos y almidón, las dos principales fuentes de reserva de carbono. La ruta TOR es inhibida por el complejo FKBP12-rapamicina (rap), y responde a nutrientes tales como nitrógeno, fósforo y CO2.
La autofagia es un mecanismo degradativo altamente conservado mediante el cual las células eucariotas eliminan y reciclan parte de su material interno para mantener la homeostasis celular y afrontar condiciones de estrés. En esta ruta catabólica, componentes celulares dañados o tóxicos son englobados en vesículas de doble membrana denominadas autofagosomas, que son liberados a la vacuola para su degradación. Nuestro grupo ha sido pionero en el estudio de este proceso catabólico en microalgas. Hemos demostrado que la autofagia es regulada negativamente por la ruta de señalización TOR y activada en respuesta a un amplio rango de condiciones de estrés tales como limitación de nutrientes, toxicidad de metales, daños en el cloroplasto o estrés oxidativo (revisado en Mallén-Ponce et al. Free Radic Bio Med 2023). Además, hemos encontrado una fuerte correlación entre un aumento en la producción de ROS y la activación de la autofagia en la mayoría de esas condiciones de estrés (Pérez-Pérez et al. Plant Physiol 2012). Nuestros resultados ponen de manifiesto que el sistema de lipidación de la proteína ATG8, que juega un papel fundamental en la formación del autofagosoma, es una diana de ROS. En primer lugar, hemos descrito el mecanismo molecular responsable de la inhibición reversible causada por ROS de la cys-proteasa ATG4 (Pérez-Pérez et al. Autophagy 2014; Pérez-Pérez et al. Plant Physiol 2016). Más recientemente, hemos demostrado que la actividad de la enzima activante de tipo E2 ATG3 está también controlada por los niveles de ROS para garantizar la correcta lipidación de ATG8 y la progresión de la autofagia en condiciones de estrés oxidativo (Mallén-Ponce et al. Plant Physiol 2024). Actualmente, estamos investigando el papel de la autofagia y de la producción de ROS en el control del metabolismo del carbono en células de Chlamydomonas sujetas a distintas fuentes nutricionales o de luz.
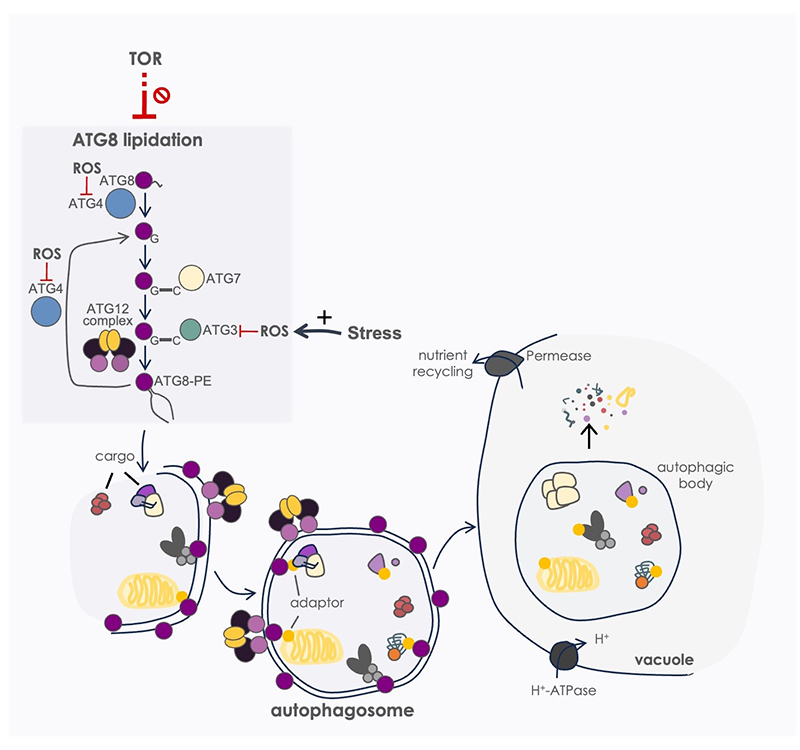
Figura 2. Autofagia en Chlamydomonas. Los componentes del sistema ATG8 (ATG3, ATG4, ATG7 y ATG8) actúan de forma coordinada para promover la biogénesis del autofagosoma. La conjugación de ATG8 al fosfolípido de membrana fosfatidiletanolamina (PE) es un paso clave tanto para la formación del autofagosoma como para la selección del cargo. Para la lipidación de ATG8, en primer lugar, ATG8 recién sintetizada es procesada por la cys-proteasa ATG4 en una Gly altamente conservada de su extremo carboxilo terminal. Posteriormente, por la acción secuencial de las enzimas ATG7 y ATG3, ATG8 se une a PE, dando lugar al complejo ATG8-PE. El complejo ATG5-ATG12-ATG16 potencia la formación de ATG8-PE. Además de procesar ATG8, ATG4 presenta una actividad delipidante, que actúa sobre ATG8-PE y libera ATG8 de las membranas del autofagosoma. El contenido del autofagosoma es degradado y reciclado en la vacuola. La autofagia es negativamente regulada por TOR y activada por condiciones de estrés que generan ROS. Dos de las principales proteínas reguladoras de autofagia, ATG4 y ATG3, son reversiblemente inhibidas por ROS.
Nuestro grupo también ha iniciado una nueva línea de investigación centrada en el estudio de la ruta de señalización TOR y la autofagia en microalgas extremófilas, ya que estos organismos han adaptado su crecimiento a condiciones ambientales extremas. Con este fin, hemos aislado una nueva microalga extremófila, Chlamydomonas urium, del nacimiento del río Tinto (Nerva), un río extremadamente ácido localizado en el sur de España. Hemos demostrado que C. urium tiene una alta tasa fotosintética, así como una alta capacidad de reciclaje de cuerpos lipidícos a través del proceso de autofagia selectiva conocido como lipofagia (Pérez-Pérez et al. New Phytologist 2024).

| Nombre | Apellidos | Categoria | Teléfonos | |
|---|---|---|---|---|
| José Luis | Crespo González | Investigador Científico CSIC | ext. 446074 | |
| Samuel | Gámez Arcas | Investigador postdoctoral | ext. 446061 | |
| Manuel Jesús | Mallén Ponce | Investigador postdoctoral | ext. 446069 | |
| Irene | Muñoz Palacios | Investigadora predoctoral | ||
| Yosu | Odriozola Gil | Investigador predoctoral | ext. 446069 | |
| María Esther | Pérez Pérez | Científica Titular CSIC | ext. 446069 | |
| Andrea | Quintero Moreno | Técnico de laboratorio | ext. 446069 | |
| María del Águila | Ruiz Sola | Profesora Contratada Doctora | 954139176 ext. 446061 |
- Mallén-Ponce MJ, Quintero-Moreno AM, Gámez-Arcas S, Grossman AR, Pérez-Pérez ME, Crespo JL. Dihydroxyacetone phosphate generated in the chloroplast mediates the activation of TOR by CO2 and light. Science Advances. 2025 (in press).
- Pérez-Pérez ME, Mallén-Ponce MJ, Odriozola-Gil Y, Rubio A, Salas JJ, Martínez-Force E, Pérez-Pulido AJ, Crespo JL. Lipid turnover through lipophagy in the newly identified extremophilic green microalga Chlamydomonas urium. New Phytologist 2024. Jul;243(1):284-298. doi.org/10.1111/nph.19811.
- Mallén-Ponce MJ, Pérez-Pérez ME. Redox-mediated activation of ATG3 promotes ATG8 lipidation and autophagy progression in Chlamydomonas reinhardtii. Plant Physiol. 2023 Dec 30;194(1):359-375. doi: 10.1093/plphys/kiad520.
- Mallén-Ponce MJ, Pérez-Pérez ME, Crespo JL. Photosynthetic assimilation of CO2 regulates TOR activity. Proc Natl Acad Sci U S A. 2022 Jan 11;119(2):e2115261119. doi: 10.1073/pnas.2115261119.
- Couso I, Pérez-Pérez ME, Ford MM, Martínez-Force E, Hicks LM, Umen JG, Crespo JL. Phosphorus Availability Regulates TORC1 Signaling via LST8 in Chlamydomonas. Plant Cell. 2020 Jan;32(1):69-80. doi: 10.1105/tpc.19.00179.
- Heredia-Martínez LG, Andrés-Garrido A, Martínez-Force E, Pérez-Pérez ME, Crespo JL. Chloroplast Damage Induced by the Inhibition of Fatty Acid Synthesis Triggers Autophagy in Chlamydomonas. Plant Physiol. 2018 Nov;178(3):1112-1129. doi: 10.1104/pp.18.00630.
- Pérez-Pérez ME, Lemaire SD, Crespo JL. Control of Autophagy in Chlamydomonas Is Mediated through Redox-Dependent Inactivation of the ATG4 Protease. Plant Physiol. 2016 Dec;172(4):2219-2234. doi: 10.1104/pp.16.01582.
- Pérez-Pérez ME, Zaffagnini M, Marchand CH, Crespo JL, Lemaire SD. The yeast autophagy protease Atg4 is regulated by thioredoxin. Autophagy. 2014;10(11):1953-64. doi: 10.4161/auto.34396.
- Pérez-Pérez ME, Couso I, Crespo JL. Carotenoid deficiency triggers autophagy in the model green alga Chlamydomonas reinhardtii. Autophagy. 2012 Mar;8(3):376-88. doi: 10.4161/auto.18864.
- Pérez-Pérez ME, Florencio FJ, Crespo JL. Inhibition of target of rapamycin signaling and stress activate autophagy in Chlamydomonas reinhardtii. Plant Physiol. 2010 Apr;152(4):1874-88. doi: 10.1104/pp.109.152520.


